RETREG1
El gen RETREG1 (regulador 1 de la reticulofagia) (Reticulophagy regulator 1 en inglés) se encuentra en el cromosoma 5 y codifica la síntesis de la proteína RETREG1 (también conocida como FAM134B o JK1 ), la cual juega un papel importante en el proceso celular de la autofagia.
Este proceso es utilizado por las células para reciclar orgánulos, células infectadas o partes celulares desgastadas o innecesarias. También descompone proteínas una vez ya han cumplido con su función.[1]
Concretamente, la proteína RETREG1 dirige la autofagia del retículo endoplasmático (RE), que procesa y transporta proteínas. La RETREG1 hace posible que la célula elimine partes del RE innecesarias y que degrade proteínas del RE excedentes o anormales. También es importante en la organización del aparato de Golgi, que distribuye las proteínas por la célula (sorting).
Esta proteína se encuentra en las neuronas sensoriales, que transmiten sensaciones de dolor, tacto y temperatura, y también en las neuronas autónomas, responsables de controlar funciones involuntarias del cuerpo, como por ejemplo la frecuencia cardíaca o la presión arterial.[2]
| Retioculophagy regulator 1 RETREG1 | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | Buscar ortólogos: PDBe, RCSB | |||
| Identificadores | ||||
| Identificadores externos | ||||
| Locus | Cr. 5 p15.1 | |||
| Estructura/Función proteica | ||||
| Tamaño | 497 (aminoácidos) | |||
| Peso molecular | 57.000 (Da) | |||
| Funciones | Regulación de la retículo-fagia, un proceso de autofagia selectiva que degrada el retículo endoplasmático (RE). Contribuye a la homeostasis celular y en la calidad del RE. | |||
| Datos biotecnológicos/médicos | ||||
| Enfermedades | Mutaciones en el gen RETREG1/FAM134B están asociadas con neuropatía sensorial hereditaria tipo II (HSAN II) y con potenciales desórdenes en enfermedades neurodegenerativas debido a su rol en el manejo del estrés celular. | |||
| Interacciones moleculares | RETREG1 interactúa con proteínas relacionadas con la autofagia como LC3, y posiblemente con proteínas de otras vías celulares relacionadas con la degradación del RE. | |||
| UniProt |
| |||
Su disfunción se asocia con enfermedades neurodegenerativas y con cáncer. En concreto, la RETREG1 desempeña una doble función en el cáncer (dependiendo del tipo de cáncer): como oncogén y como supresor tumoral, ya que puede regular la apoptosis de las células tumorales mediante la reticulofagia.
Genética y Síntesis
editarEstructura génica de RETREG1
editarLa estructura de este gen incluye un promotor, cinco exones y cuatro intrones, abarcando aproximadamente 6.5 kb de ADN. La presencia de múltiples exones permite la generación de isoformas de ARNm a través del splicing alternativo, un fenómeno que confiere flexibilidad funcional y regulativa a la proteína resultante.
Componentes estructurales
editarPromotor:
editarEl promotor de RETREG1 es una región proximal al gen a la que se unen proteínas relevantes para iniciar la transcripción. En general los promotores contienen secuencias específicas donde se unen los factores de transcripción y la ARN polimerasa II. Dentro de este promotor, se pueden encontrar elementos específicos como:
- Elemento TATA (TATA box): Reconocido por el complejo de preiniciación de la ARN polimerasa, en particular por el factor de transcripción TFIID.
- Elementos de respuesta a citoquinas: Como los sitios de unión de NF-kB y AP-1, que son cruciales para la activación en respuesta a señales inflamatorias y de estrés.
- Elementos de respuesta a factores de crecimiento: Como los sitios de unión para C/EBP (CCAAT/enhancer-binding protein), que regulan la expresión del gen en función del estado metabólico de la célula.
Exones e intrones
editarEl gen RETREG1 contiene cinco exones:
- RETREG1-Exón1: Inicia la codificación de la proteína y contiene la secuencia del péptido señal que dirige la proteína hacia el RE.
- RETREG1-Exón2: Contribuye a la región de unión a sustratos. Este exón está involucrado en la unión a proteínas y lípidos, en particular, puede interactuar con proteínas como el receptor RET, lo que sugiere un papel en la señalización celular.
- RETREG1-Exón3: Involucrado en la actividad enzimática de la proteína. Este exón contiene residuos de aminoácidos que son importantes para la actividad catalítica de RETREG1.
- RETREG1-Exón4: Este exón contiene motivos que pueden ser reconocidos por ubiquitina ligasas, como Mdm2 o E3, que son responsables de marcar la proteína para su degradación proteasomal, regulando así su concentración en la célula.
- RETREG1-Exón5: Este exón codifica para dominios que permiten la interacción con varias proteínas:
- HSP90 (Heat Shock Protein 90): Una chaperona que ayuda en la correcta conformación de proteínas y en la estabilización de proteínas recién sintetizadas.
- HSP70 (Heat Shock Protein 70): Otra chaperona que colabora en el plegamiento correcto y la prevención de la agregación de proteínas.
- Cofilina: Participa en la remodelación del citoesqueleto, lo que sugiere que RETREG1 podría tener un papel en la dinámica celular y la migración.
RETREG1 también posee cuatro intrones que son segmentos no codificantes. Los intrones son eliminados del pre-ARNm durante el procesamiento del ARN, permitiendo la unión de los exones mediante el splicing alternativo. Esto permite que se generen múltiples isoformas de ARNm, lo que proporciona diversidad funcional a la proteína resultante.
Elementos reguladores: La región promotora y los intrones pueden contener enhancers (potenciadores) y silencers (silenciadores), que son secuencias que modulan la actividad transcripcional.
La capacidad de RETREG1 para ser regulado por múltiples señales es esencial para la homeostasis celular y la adaptación a cambios en el entorno. Los factores de transcripción, junto con modificaciones epigenéticas, como la metilación del ADN, afectan la expresión de este gen. Por ejemplo:
- Modificaciones de histonas: Las modificaciones en las histonas, como la acetilación mediada por proteínas como CBP/p300, pueden abrir la cromatina y facilitar el acceso de los factores de transcripción al promotor de RETREG1.
- Mecanismos de respuesta: En respuesta a estrés celular o condiciones inflamatorias, la activación de la vía de NF-κB puede inducir la transcripción de RETREG1, destacando su papel en la respuesta inmune.[3]
Transcripción y procesamiento del ARN de RETREG1
editarEl gen RETREG1 codifica para una proteína involucrada en el tráfico y degradación de proteínas en el RE. Además de participar en la autofagia del RE (ER-phagy), esta proteína juega un rol crítico en el mantenimiento de la homeostasis celular y se ha asociado con la degradación selectiva de fragmentos del RE mediante el sistema lisosomal.[4]
Variables de splicing
editarEl proceso de splicing alternativo permite que un solo gen codifique para múltiples isoformas de una proteína al eliminar intrones y empalmar exones de distintas formas. En el caso de RETREG1, se han identificado varias isoformas generadas por splicing alternativo, las cuales pueden diferir en sus dominios funcionales y, por lo tanto, en sus funciones celulares.[5]
- Mecanismos de splicing en RETRE1
El splicing alternativo de RETREG1 es regulado por un conjunto de proteínas de unión al ARN (RBPs, RNA-binding proteins) y factores de splicing que determinan la selección de sitios específicos de corte y empalme. Entre estos factores destacan:
- SF3B1 (Subunidad B1 del Factor de Splicing 3b): Es un componente del complejo de corte y empalme (spliceosome) que se une a las secuencias de empalme en el intrón y facilita la eliminación de intrones.[6]
- SRSF (Proteínas de la familia de Serina/Arginina): Estas proteínas, especialmente SRSF1 y SRSF3, se unen a secuencia específicas en el ARNm de RETREG1 y regulan el empalme de exones específicos, promoviendo o inhibiendo la inclusión de ciertos exones.
- hnRNPs (Proteínas Ribonucleoproteicas Nucleares Heterogéneas): Las proteínas hnRNP, como hnRNP A1 y hnRNP C, también modulan el splicing alternativo y pueden competir con las proteínas SRSF por sitios de unión en el ARNm de RETREG1, afectando la selección de exones.
- Isoformas de RETREG1 generadas por splicing
Se han identificado varias isoformas de RETREG1 mediante análisis de ARN y secuenciación. Estas isoformas pueden variar en función de su ubicación subcelular y en su habilidad para interactuar con otras proteínas, lo que sugiere que el splicing alternativo podría regular la función de RETREG1 en distintos contextos celulares.
- Isoformas (completa): Contiene todos los exones y es funcionalmente activa en el proceso de autofagia del RE.
- Isoformas B (incompleta): Excluye ciertos exones que codifican para dominios de interacción específicos, lo cual podría limitar su capacidad para participar en la degradación del RE.
Regulación génica y factores de transcripción involucrados
editarLa expresión del gen RETREG1 es un proceso complejo regulado por diversos factores de transcripción y elementos cis-reguladores presentes en la región promotora y en los intrones. Estos factores de transcripción responden a estímulos celulares y ambientales, ajustando la expresión de RETREG1 según las necesidades fisiológicas de la célula.[7]
- Factores de transcripción clave en la regulación de RETREG1
La expresión de RETREG1 está influenciada por factores de transcripción que regulan genes implicados en la respuesta al estrés del RE, la autofagia y el tráfico intracelular. Entre los factores más relevantes destacan:
- ATF6 (Factor de Transcripción Activador 6): Como parte de la respuesta al estrés del RE (UPR, por sus siglas en inglés, Unfolded Protein Response), ATF6 se activa en condiciones de acumulación de proteínas mal plegadas en el RE. ATF6 puede unirse a la región promotora de RETREG1 y aumentar su transcripción para promover la autofagia del RE y aliviar el estrés celular.[8]
- XBP1 (Proteína de Unión a la Caja X1): Otro factor clave en la UPR. XBP1 se activa mediante splicing en respuesta al estrés del RE, lo que permite unirse a elementos reguladores en el ADN de RETREG1. La activación de XBP1 conduce a la transcripción de RETREG1 y de otros genes involucrados en la degradación y renovación de proteínas del RE.[9]
- TFEB (Factor de Transcripción EB): TFEB es un regulador maestro de genes involucrados en la autofagia y la biogénesis lisosomal. Bajo condiciones de estrés o necesidad de autofagia, TFEB se transloca al núcleo y activa genes relacionados con la degradación lisosomal y la autofagia del RE, incluyendo RETREG1.[10]
- NRF1 (Factor Nuclear Eritroide 2-Relacionado 1): Este factor de transcripción responde al estrés oxidativo y regula la expresión de genes implicados en la reparación y mantenimiento celular, entre ellos RETREG1. NRF1 puede inducir la transcripción de RETREG1 en respuesta a señales de daño celular y estrés oxidativo.[10]
- Coactivadores y represores en la regulación de RETREG1
Además de los factores de transcripción mencionados, existen coactivadores y represores que modulan la actividad de estos factores en la región promotora de RETREG1:
- P300/CBP: Estos coactivadores interactúan con ATF6 y otros factores de la UPR para amplificar la transcripción de RETREG1. Actúan como acetiltransferasas de histonas, relajando la cromatina y permitiendo un acceso más eficiente de los factores de transcripción.[11]
- HDACs (Desacetilasas de Histonas): Las HDAC pueden actuar como represores al compactar la cromatina, reduciendo la accesibilidad de los factores de transcripción a la región promotora de RETREG1. En condiciones de estrés o necesidad de autofagia, la inhibición de HDACs favorece la transcripción de RETREG1.[12]
- Elementos cis-reguladores y potenciadores
La región promotora de RETREG1 contiene varios elementos cis-reguladores, como el elemento de respuesta al estrés del RE (ERSE, Endoplasmatic Reticulum Stress Response Element) y el elemento de respuesta a antioxidantes (ARE, Antioxidant Response Element). Estos elementos permiten la unión de factores como ATF6 y NRF1 bajo condiciones específicas. Adicionalmente, se han identificado potenciadores (enhancers) en regiones intrónicas que contribuyen a la regulación a largo plazo de la expresión de RETREG1.[13]
Síntesis y traducción de la proteína RETREG1
editarLa síntesis y correcta traducción de RETREG1 son pasos claves que requieren una regulación precisa y la participación de chaperonas para asegurar su adecuada función.
Regulación de la síntesis de la proteína
editarLa regulación de la síntesis de RETREG1 incluye factores de iniciación de la traducción, modificaciones postranscripcionales del ARNm, y mecanismos de control de calidad para evitar la traducción de proteínas mal plegadas.[14]
- Factores de iniciación de la traducción
Este proceso inicia en los ribosomas, donde varios factores de iniciación actúan para regular el inicio de la síntesis de proteínas:
- eIF2-α (Factor de Iniciación de la Traducción eucariota 2-α): Cuando el RE está bajo estrés, eIF2-α puede sufrir fosforilación. Este proceso disminuye la traducción general de proteínas en la célula, permitiendo que se produzca un conjunto específico de proteínas, entre ellas RETREG1. Esto garantiza que la célula enfoque sus recursos en la producción de proteínas que ayudan a aliviar el estrés y mantener el equilibrio celular.[14]
- eIF4E (Factor de Iniciación de la Traducción eucariota 4E): Este factor desempeña un papel crucial en la regulación de la traducción de ARNm específicos, especialmente aquellos implicados en la autofagia y el control de calidad en el RE. La regulación de eIF4E ajusta la síntesis de RETREG1 en función de las señales de crecimiento y las condiciones de estrés celular.[15]
- Regulación mediante microARNs (miARNs)
Los microARNs son pequeñas moléculas de ARN no codificante que pueden unirse a secuencias específicas en el ARNm de RETREG1, bloqueando su traducción o promoviendo su degradación.[16]
- miR-29a y miR-124: Estos microARNs se han relacionado con la regulación de genes involucrados en la autofagia y el tráfico de proteínas. La unión de estos microARNs al ARNm de RETREG1 podría inhibir su traducción en condiciones en que la célula necesita reducir la autofagia del RE, como en estados de menor demanda de degradación de proteínas.[16][17]
- Regulación postranscripcional y modificaciones del ARNm
Además de la regulación mediante microARNs, el ARNm de RETREG1 sufre modificaciones químicas que afectan a su estabilidad y traducción.
- Metilación de N6-metiladenosina (m6A): La adición de m6A en el ARNm de RETREG1 es una modificación que puede incrementar la estabilidad del ARNm o facilitar su traducción. Esta modificación suele ser reconocida por proteínas específicas, como YTHDF1, que pueden promover la síntesis de RETREG1 en respuesta a señales de estrés celular.[18]
- Poliadenilación: La extensión de la cola de poliadenina (poli-A) en el extremo 3' del ARNm de RETREG1 afecta su estabilidad y la eficiencia de su traducción. Una mayor poliadenilación puede aumentar la estabilidad del ARNm, permitiendo una síntesis continua de RETREG1 durante la respuesta al estrés del RE.[19]
Interacciones de chaperonas en la síntesis de RETREG1
editarUna vez sintetizada, la proteína RETREG1 requiere un correcto plegamiento para la realización de su función en la autofagia del RE. Una serie de chaperonas moleculares facilitan este proceso, ya que ayudan a prevenir el plegamiento erróneo y la agregación de la proteína, además de asegurarse de que RETREG1 alcance su estructura nativa funcional antes de ser transportada al RE y desempeñar su función.
- Chaperonas del RE
Algunas chaperonas implicadas en el correcto plegamiento de RETREG1 incluyen:
- BiP/GRP78 (Binding Immunoglobulin Protein/Glucose-Regulated Protein 78): BiP es una chaperona clave en el RE que se une a RETREG1 recién sintetizado para ayudar en su plegamiento y prevenir la formación de agregados. BiP reconoce proteínas mal plegadas y actúa como una primera línea de control de calidad en el RE, asegurándose de que solo las proteínas correctamente plegadas pasen al siguiente paso de procesamiento.[20][21][22]
- Calnexina y Calreticulina: Estas chaperonas específicas del RE se unen a glucoproteínas en fase temprana de plegamiento. Como RETREG1 contiene sitios de glucosilación, la calnexina y la calreticulina pueden interactuar temporalmente con esta proteína durante su plegado para asegurar que adquiera la conformación estructural adecuada.[23][24][25]
- Control de calidad y degradación de proteínas mal plegadas
ERAD (Endoplasmatic Reticulum-Associated Degradation) es el sistema de control de calidad del RE que asegura que, en caso de que RETREG1 no logre plegarse correctamente, esta proteína sea marcada para su degradación. [26][27][28]
- Proteínas de ERAD: Componentes del sistema ERAD, como HRD1 y SEL1L, reconocen proteínas mal plegadas en el RE y las marcan para su retrotranslocación al citoplasma, donde serán ubiquitinadas y posteriormente degradadas por el proteasoma. Si RETREG1 no logra adoptar su estructura nativa, es canalizada a través de esta vía para prevenir efectos adversos en la célula.[26][29][30][31]
Este mecanismo de control de calidad evita la acumulación de proteínas mal plegadas en el RE y permite la síntesis de nuevas proteínas funcionales.
Estructura Molecular
editarLa proteína RETREG1 cuenta con 497 aminoácidos.[32] Está formada por un dominio en C-terminal estructurado en forma de hélice y por un extremo N-terminal que contiene dos segmentos hidrofóbicos con 35 aminoácidos cada uno.[33] Estos dos segmentos se encuentran separados por un loop hidrofílico de 60 aminoácidos, el cual forma una estructura similar a otras proteínas del RE que llevan a cabo la misma función que RETREG1.[32]
Dominios funcionales de la proteína
editarLa proteína cuenta con dos estructuras principales: el motivo LC3-interacting region (LIR) y el dominio reticulon-homology domain (RHD). Los dos emplean un papel imprescindible en la correcta funcionalidad de la proteína, pero no son los únicos elementos que se distinguen en esta, ya que en su estructura también se encuentra un dominio parecido al Factor de Crecimiento Epidérmico (EGF), 3 puntos de N-glucosilación, 3 puntos de miristoilación[34] y diferentes puntos de fosforilación.
Dominio de interacción con el RE
editarEl dominio RHD contiene entre 80 y 260 residuos. Se mantiene al lado de los extremos N-terminal y C-terminal, ambos citoplasmáticos, por la presencia de una región intrínsecamente desordenada (IDPR).[33] La IDPR de C-terminal de RETREG1 está unida a LIR, a través de la cual la proteína interacciona con LC3. Aparte, la IDPR une el RE, que se degradará posteriormente, a la maquinaria relativa a la autofagia. Independientemente, la IDPR influencia en la forma curvada de la membrana, pero la unión de esta con otros elementos especializados en la curvatura de la membrana (como el dominio RHD) provoca una aceleración del proceso y una mayor eficacia en la creación de vesículas.[35] La función de la IDPR de N-terminal es desconocida.[33]
El dominio RHD contiene seis partes fundamentales: cuatro hélices transmembrana (TM1, TM2, TM3 y TM4) y dos hélices anfipáticas citoplasmáticas (AHL y AHC).[36]
A partir de simulaciones moleculares se ha probado que estos segmentos transmembrana se pliegan en dos grupos de horquillas, cada una de las cuales cuenta con un loop polar de 3-4 residuos. Un loop conecta TM1 con TM2, y el otro mantiene TM3 ligado a TM4. La estructura TM1-TM2 está conectada con TM3-TM4 a partir de AHL, y el AHC se une con el extremo C-terminal citoplasmático del dominio RHD. Las hélices AHL y AHC cuentan con regiones hidrofóbicas ancoradas en la bicapa lipídica de la membrana del RE.
Las hélices transmembrana se posicionan ligeramente inclinadas, por lo que se induce a su interacción con los loops del lumen del RE.
El conjunto de la estructura comentada anteriormente (que incluye los segmentos transmembrana y las hélices) toma forma asimétrica, lo que conduce a la curvatura de la bicapa lipídica, que ya se ha mencionado anteriormente.[36]
Es fundamental destacar los escasos conocimientos que la comunidad científica posee respecto a la estructura tridimensional del dominio RHD. Por lo tanto, los mecanismos de acción a partir de los cuales el dominio RHD influye en la forma de la membrana del RE son, en gran medida, desconocidos.[33]
El dominio RHD también contiene tres regiones de fosforilación (S149, S151 y S153), todas ellas involucradas en la oligomerización de RETREG1.[36]
Cabe destacar que no se trata de un dominio exclusivo de RETREG1, ya que también se encuentra en otras proteínas que cumplen una función similar a esta como, por ejemplo, SEC62, RTN3L, CCPG1, ATL3 o TEX264. Por lo tanto, el dominio RHD es fundamental para la ejecución de la reticulofagia.[37]
Motivos y regiones críticas para la autofagia selectiva
editarEl motivo LIR (residuos 453–458 en C-terminal) presenta receptores participantes en la autofagia selectiva. Su función se basa en unir la proteína RETREG1 con proteínas de la familia LC3/GABARAP, las cuales se caracterizan por su función modificadora de la autofagia. Se han detectado ciertas mutaciones en este motivo que inducen a la aparición de enfermedades, muchas de las cuales se relacionan con neuropatías e infecciones víricas.[36] Otra situación crítica se produce frente a la ausencia del motivo LIR, la cual no permite la fragmentación del RE.[33]En estas situaciones, no se induce a la reticulofagia.
Cabe destacar que en la autofagia se distinguen otros componentes bioquímicos, sobre todo reguladores de este proceso. Destacamos el ATG5 y BECN1, los cuales son esenciales para la fragmentación del RE y su posterior degradación. Por lo tanto, la función de inducir la reticulofagia no solamente es dependiente de RETREG1.[36]
Variantes estructurales
editarDistinguimos diferentes parálogos de RETREG1, o lo que es lo mismo, de FAM134B. Algunos ejemplos serían FAM134A o FAM143C. Estos se diferencian entre sí a partir de la longitud del extremo C-terminal. Todos ellos mantienen ciertas estructuras iguales, como el dominio LIR o las horquillas TM, pero se destaca un 30% de secuencias que varían entre parálogos. Las diferencias principales se encuentran en la distribución del cargo y en la forma de las vesículas a las que dan lugar; la vesícula formada a partir de FAM134A es mucho más compacta que las vesicales formadas a partir de FAM134B o de FAM134C.
La función propia de la proteína se mantiene en todas sus variantes estructurales, ya que FAM134A y FAM134C también inducen a la fragmentación del RE, dependiendo siempre del dominio LIR, el cual también está presente en FAM134B. Cabe destacar que todas las variantes estructurales requieren de modificaciones post-traduccionales para su correcta funcionalidad.
Por otro lado, su regulación difiere entre parálogos; FAM134A y FAM134C se encuentran en estado inactivo en condiciones basales; para ser activados se requiere de una senyal externa. FAM134B, en un organismo sano, se encuentra en estado activo sin necesidad de una señalización previa.[38]
Función Biológica
editarLa proteína RETREG1 es una reguladora de procesos autofagocíticos en distintos orgánulos de la célula y desempeña un papel importante en la degradación de diversas moléculas en el RE y en los autofagosomas. Además, puede actuar como una proteína en el cis-Golgi (CG), pero todavía se desconoce su función exacta en este orgánulo. También tiene cierta relación con los lisosomas y las mitocondrias. Principalmente, es uno de los receptores más importantes en la regulación de la autofagia del RE, un proceso conocido como reticulofagia (o ER-phagy).[36]
Relación RETREG1 con el RE
editarHomeostasis del RE y la supervivencia celular
editarLa homeostasis del RE se mantiene gracias a la reticulofagia, ya que consiste en una degradación selectiva de ciertos subdominios del orgánulo. La degradación puede deberse a varios factores, como el mal plegamiento de proteínas, que genera un estrés en el RE, o a un subdominio dañado o innecesario que compromete la supervivencia de la célula. Por tanto, RETREG1, al ser uno de los receptores encargados de regular la reticulofagia, desarrolla un papel distintivo en el reconocimiento y marcaje de subdominios RE específicos para su degradación.[39]
Sin embargo, la reticulofagia podría desempeñar una función doble en la supervivencia celular. Aunque la reticulofagia ayuda a restaurar la homeostasis del RE mediante la eliminación de subdominios o proteínas mal plegadas, un exceso de autofagia del RE podría inducir la muerte celular por apoptosis. En investigaciones recientes, se ha observado como una mutación en RETREG1 por la introducción de G216R en el RHD incrementa excesivamente la reticulofagia y desencadena la muerte celular de neuronas sensoriales. Por tanto, se ha postulado que RETREG1 es un regulador crucial dentro de la célula.[36]
Mecanismos de acción y señalización de RETREG1 en la reticulofagia
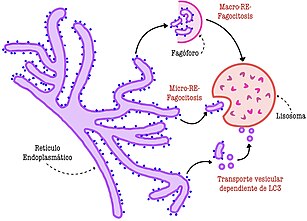
editarCuando hay una sobrecarga de calcio o estrés oxidativo en la célula, se genera un estrés dentro del RE que activa la reticulofagia. Durante la reticulofagia, las membranas del RE se deforman y forman vesículas que posteriormente son engullidas por un autofagosoma[36], una vesícula con membrana cerrada constituida a partir de una bicapa lipídica, que contiene en su interior las moléculas que la célula eliminará durante la autofagia.[33] RETREG1-RHD regula la deformación de la membrana del RE en el primer paso de la degradación.[36]
RETREG1 es el primer receptor en la degradación del RE.[39]La sobrecarga de calcio activa la quinasa CAMK2B, la cual induce la reticulofagia mediante la fosforilación de RETREG1 en la serina 151. La fosforilación es esencial para la oligomerización de RETREG1 y la fragmentación del RE.[36]
Simultáneamente, la enzima USP20 desubicuitiniza RETREG1 al cortar las cadenas de ubicuitina K48 y K63, las cuales estaban anteriormente unidas a la proteína. Las desubicuitinación estabiliza RETREG1 para que pueda desempeñar su papel en la reticulofagia.[39]
Tras la señalización, RETREG1-RHD induce una curvatura de la membrana del RE mediante las horquillas TM y hélices anfipáticas (AHs) localizados en el RHD. Las horquillas son elementos cruciales para el anclaje estable en la membrana y las AHs asisten en la interacción de RETREG1 con la bicapa lipídica del RE y la curvatura de la membrana. Además, ambos contribuyen al agrupamiento de otros RETREG1-RHD, lo cual aumenta la deformación de la membrana.
Al mismo tiempo, el motivo LIR en la C-terminal de RETREG1 reconoce la membrana autofagosomal, uniéndose directamente al LC3 del fagóforo y generando una fuerza de arrastre. A consecuencia de esta fuerza, la membrana del RE forma vesículas que se acaban cerrando sobre ellas mismas y se desprenden del RE para ser envueltas por el autofagosoma. Este último se fusiona con un lisosoma, un organelo con un pH más ácido al contener enzimas (hidrolasas lisosomales ácidas) especializadas en la degradación de moléculas. Por tanto, después de la fusión de los dos compartimentos celulares, se efectúa la degradación completa del material del RE dentro del lisosoma.[36]
Relación con lisosomas y mitocondrias
editarLa desregulación o sobrecarga de Ca2+ en el RE genera una liberación de iones de calcio que son transportados desde el RE, a través de las mitochondria-associated membranes (MAMs), hasta las mitocondrias. Este transporte induce una sobrecarga de Ca2+ en la mitocondria, la cual altera la homeostasis. Como consecuencia, la mitocondria no desempeña su función correctamente y se activa la autofagia mitocondrial por apoptosis para eliminar el organelo. La autofagia mitocondrial sigue un proceso muy similar a la reticulofagia, ya que la mitocondria también es envuelta por un fagosoma y se acaba degradando en el lisosoma. Sin embargo, se ha observado que la reticulofagia mediada por RETREG1 es capaz de atenuar la apoptosis en las mitocondrias de neuronas del hipocampo mediante la regulación de intercambio de Ca2+ entre el RE y la mitocondria. Por tanto, es probable que exista una relación entre ambos organelos crucial para la supervivencia de la célula.
De forma similar, una sobrecarga de Ca2+ en el RE suele resultar en inestabilidad lisosomal y provoca la apoptosis del lisosoma. Por tanto, se cree que es posible que la reticulofagia mediada por RETREG1 mantiene la homeostasis en el lisosoma al regular el Ca2+ del RE.[40]
Relación con el aparato de Golgi
editarEl regulador RETREG1 también se encuentra en el CG. Los resultados de las investigaciones sobre la función de esta proteína en el CG demuestran que RETREG1 es capaz de inhibir la acetilación de ATG9A y fomentar su ubicuitinación. ATG9A es una proteína que, al captar la señal de estrés en el CG, se ubicuitiniza e induce la fragmentación del Golgi. La fragmentación es un origen de aumento autofagosomal que inicia la autofagia del Golgi para restablecer la homeostasis, aunque el mecanismo específico de RETREG1 en la autofagia del orgánulo se desconoce.[40]
Importancia médica
editarLa ausencia o la expresión no funcional de la proteína RETREG1 altera el recambio del RE y, por lo tanto, está involucrada diversas enfermedades humanas. Esto es debido a mutaciones en el gen RETREG1 que se heredan de manera autosómica recesiva.
La inhibición de FAM134B contribuye a perjudicar la proteostasis en el RE debido a la acumulación de proteínas mal plegadas o agregadas, lo que a su vez conduce a una supervivencia neuronal comprometida y enfermedades degenerativas neuronales progresivas, como HSANIIB, Niemann-Pick tipo C, Alzheimer, Párkinson, y otras enfermedades como cáncer.
HSANIIB En el caso de la enfermedad HSANIIB, que es un subtipo de la enfermedad HSAN, los pacientes sufren una degeneración de los nervios periféricos que provoca una pérdida sensorial grave (va desde un entumecimiento local hasta la incapacidad total de sentir dolor). En casos extremos resulta en una mutilación grave.[41]
Niemann-Pick tipo C
Por otro lado, la enfermedad de Niemann-Pick tipo C es un trastorno neurodegenrativo progresivo y fatal causado principalmente por la acumulación de colesterol no esterificado dentro de las células. Aproximadamente, el 95% de los pacientes presentan mutaciones en el gen que codifica la glucoproteina NPC1. Uno de los métodos para degradar esta mutación es la reticulofagia mediada por la proteína RETREG1. Por ello, se ha encontrado que los pacientes con Niemman-Pick tienen una proporción de reticulofagia alterada más alta que la del grupo control.[42]
Alzheimer y Párkinson
Además, se sabe que la autofagia basal es esencial para mantener la homeostasis neuronal, y que la disfunción de la autofagia está estrechamente relacionada con el Alzheimer y el Párkinson. Consecuentemente, se está investigando el efecto que tiene la reticulofagia en las enfermedades mencionadas.
Cáncer
En cuanto al cáncer, las mutaciones en la proteína RETREG1 pueden tener efectos opuestos según el tipo de cáncer: puede actuar como oncogén, ya que se ha encontrado en células escamosas tumorales del esófago y adenocarcinomas colorrectales, y a su vez como supresor tumoral en células de glioma, ya que inhibe el crecimiento de estas células cancerosas y regula la apoptosis.
Aún así, se necesitan más conocimientos para evaluar si la reticulofagia puede considerarse una terapia útil contra el cáncer.[43]
Referencias
editar- ↑ Costas, Mónica A,; María F. Rubio (2017). «Autofagia, Una Estrategia de Supervivencia Celular». Medicina (Buenos Aires) (SciELO) 774 (4): 314-320.
- ↑ «RETREG1 gene: MedlinePlus Genetics». Medlineplus.gov (en inglés). Consultado el 31 de octubre de 2024.
- ↑ Rogozin, Igor B.; Alexander V. Sverdlov; Vladimir N. Babenko; Eugene V. Koonin (2005). «Analysis of Evolution of Exon-Intron Structure of Eukaryotic Genes». Briefings in Bioinformatics (OUP Academic) 6 (2): 118-134.
- ↑ Reggiori, Fulvio; Molinari, Maurizio (2022). «Er-Phagy: Mechanisms, Regulation, and Diseases Connected to the Lysosomal Clearance of the Endoplasmic Reticulum». Physiological Reviews (REVISIÓN) 102 (3): 1393-1448.
- ↑ Vázquez Calvo, Claudia (2021). Variantes de Splicing: Detección y Su Efecto En El Mrna (Tesis). Repositori Institucional (O2): Página de inicio.
- ↑ González-Rodríguez, Patricia; Daniel J. Klionsky; Bertrand Joseph (2022). «Autophagy Regulation by RNA Alternative Splicing and Implications in Human Diseases». Nature communications (REVISIÓN) 13 (2735).
- ↑ Cinque, Laura; Chiara De Leonibus; Maria Iavazzo; Natalie Krahmer; Daniela Intartaglia; Francesco Giuseppe Salierno;; Rossella De Cegli, et al. (2020). «MiT/TFE factors control ER‐phagy via transcriptional regulation of FAM134B». The EMBO Journal 39 (17). PMC 7459426. PMID 32716134.
- ↑ Yang, Ming; Shilu Luo; Xi Wang; Chenrui Li; Jinfei Yang;; Xuejing Zhun; Li Xiao; Lin Sun (2021). «Er-Phagy: A New Regulator of ER Homeostasis». Frontiers in Cell and Developmental Biology. (Front. Cell Dev. Biol.) (REVISIÓN). Cell Death and Survival 9: 684526.
- ↑ Cherubini, Alessandro; Zito, Ester (2022). «Er Stress as a Trigger of UPR and ER-Phagy in Cancer Growth and Spread». Frontiers in oncology (Front. Oncol.) (REVISIÓN). Molecular and Cellular Oncology 12: 997235. PMC 9667062. PMID 36408145.
- ↑ Saltar a: a b Gebrie, Alemu. (2023). «Transcription Factor EB as a Key Molecular Factor in Human Health and Its Implication in Diseases». SAGE Open Medicine (REVISIÓN) 11. PMC 9986912. PMID 36891126.
- ↑ Yang, Fengguang; Daxue Zhu; Zhaoheng Wang; Yingping Ma; Liangzeng Huang; Xuewen Kang; Bing Ma (2022). «Role of Advanced Glycation End Products in Intervertebral Disc Degeneration: Mechanism and Therapeutic Potential». Oxidative Medicine and Cellular Longevity (REVISIÓN) 2022: 7299005. PMC 9789911. PMID 36573114.
- ↑ Li, Jia-Yao; Tian Tian; Bing Han; Ting Yang; Yi-Xin Guo; Jia-Yu Wu; Yu-Si Chen; Qin Yang et al. (2023). «Suberoylanilide Hydroxamic Acid Upregulates Reticulophagy Receptor Expression and Promotes Cell Death in Hepatocellular Carcinoma Cells». World Journal of Gastroenterology 29 (34): 5038-5053.
- ↑ Man, Zhang; Zhangshun Wang; Qing Zhao; Qian Yang; Cuiwei Yang; Yanfen Liu (2023). «USP20 Deubiquitinates and Stabilizes the ER-Phagy Receptor FAM134B to Drive ER-Phagy». Autophagy (bioRxiv) 20 (8): 1780-1797.
- ↑ Saltar a: a b Peláez, Fernando (1989). «Regulación de La Síntesis de Proteínas a Nivel Del Factor de Iniciación Eif-2 En Células Eucarióticas». Dialnet (Tesis).
- ↑ Castro Sanz, Ana B. (1970). «Estudio de La Regulación Del Factor de Iniciación de La Traducción Eif4e Mediante Su Interacción Con La Proteína Ceres». Dialnet (Tesis).
- ↑ Saltar a: a b Sun, Xu; Yanpeng Yang; Xiaona Meng; Jia Li; Xiaoli Liu; Huaimin Liu (2023). «Panoptosis: Mechanisms, Biology, and Role in Disease». Immunological Reviews (REVISIÓN) 321 (1): 246-262. (requiere suscripción).
- ↑ Conley, Jessica; Benjamin Genenger; Bruce Ashford; Marie Anson (2024). «miRNA Dysregulation in Keratinocyte Carcinomas: Clinical Evidence, Experimental Validation, and Future Directions». Int J Mol Sci (REVISIÓN) 25 (15): 8493.
- ↑ Martínez García, Fernando (2014). Análisis de La Dinámica de Replicación de Un Virus de Rna de Plantas y Del Uso de Micrornas Artificiales Como Estrategia Antiviral (Tesis). RIUNET.
- ↑ Gayubas Balaguer, Beatriz (2023). Función de las Proteínas VQ1 y VQ10 Codificadas por Genes Inducibles por Hipoxia, Óxido Nítrico, y Estrés Oxidativo en la Regulación del Desarrollo y las Respuestas a Estrés en Arabidopsis Thaliana (Tesis). RIUNET.
- ↑ Luo, Rongjin; Huaizhen Liang; Weifeng Zhang; Gaocai Li; Kangcheng Zhao; Wenbin Hua; Yu Song; Cao Yang (2022). «Retreg1-Mediated ER-Phagy Activation Induced by Glucose Deprivation Alleviates Nucleus Pulposus Cell Damage via ER Stress Pathway». SciEngine 54 (4): 524-536.
- ↑ Ibrahim M. Ibrahim; Doaa H. Abdelmalek; Abdo A. Elfiky (2019). «Grp78: A Cell’s Response to Stress». Life Sciences (REVISIÓN) 226: 156-163.
- ↑ Morris, Jill A.; Andrew J. Dorner; Chris A. Edwards; Linda M. Hendershot; Randal J. Kaufman (1996). «Immunoglobulin Binding Protein (Bip) Function Is Required to Protect Cells from Endoplasmic Reticulum Stress but Is Not Required for the Secretion of Selective Proteins». Journal of Biological Chemistry 272 (7): 4327-4334.
- ↑ Zhang, Jing; Bin Wang; Xiaoxiao Gao; Cheng Peng; Chao Shan; Silas F. Johnson; Richard C. Schwartz; Yong-Hui Zheng (2022). «RNF185 Regulates Proteostasis in Ebolavirus Infection by Crosstalk between the Calnexin Cycle, Erad, and Reticulophagy». Nature Communications 13 (1): 6007.
- ↑ Caramelo, Julio J.; Parodi, Armando J. (2008). «Getting in and out from Calnexin/Calreticulin Cycles». THE JOURNAL OF BIOLOGICAL CHEMISTRY (REVISIÓN) 283 (16p): 10221-10225.
- ↑ Muller-Taubenberger, A. (2001). «Calreticulin and Calnexin in the Endoplasmic Reticulum Are Important for Phagocytosis». The EMBO Journal 20 (23): 6772-6782.
- ↑ Saltar a: a b Buchberger, Alexander; Bukau, Bernd; Sommer, Thomas (2010). «Protein quality control in the cytosol and the endoplasmic reticulum: brothers in arms». Mol Cell (REVISIÓN) 40 (2): 238-252. PMID 20965419.
- ↑ Morens, David M.; Fauci, Anthony S. (2020). «Emerging Pandemic Diseases: How We Got to COVID-19». Cell Press (REVISIÓN) 182 (5): 1077-1092. PMC 7428724. PMID 32846157.
- ↑ Shi, Wembo; Ran Ding; Yilin Chen; Fubo Ji; Junfang Ji; Weirui Ma; Jianping Jin (2024). «The HRD1-SEL1L ubiquitin ligase regulates stress granule homeostasis in couple with distinctive signaling branches of ER stress». iScience 27 (7): 110196. PMC 11228786. PMID 38979013.
- ↑ Koenig, Paul-Albert; Hidde L Ploegh. (2014). «Protein Quality Control in the Endoplasmic Reticulum». F1000prime reports 6 (49).
- ↑ Wei, Xiaoqiong (2024). «Proteomic Screens of SEL1L-HRD1 ER-Asso- Ciated Degradation Substrates Reveal Its Role in Glycosylphosphatidylinositol-Anchored Protein Biogenesis.». Nature Communications 15 (659).
- ↑ Shi, Wembo; Ran Ding; Yilin Chen; Fubo Ji; Junfang Ji; Weirui Ma; Jianping Ji (2024). «The Hrd1-SEL1L Ubiquitin Ligase Regulates Stress Granule». iScience 27 (7): 110196. doi:10.1016/j.isci.2024.110196.
- ↑ Saltar a: a b «613114 - Reticulophagy Regulator 1; Retreg1». OMIM.
- ↑ Saltar a: a b c d e f Popelka, Hana; Daniel J. Klionsky (2020). «Molecular Dynamics Simulations Reveal How the Reticulon-Homology Domain of the Autophagy Receptor RETREG1/FAM134B Remodels Membranes for Efficient Selective Reticulophagy». Autophagy (Taylor & Francis Online) 16 (4): 585-588.
- ↑ Udenwobele, Daniel Ikenna; Ruey-Chyi Su; Sara V Good; Terry Blake Ball; Shailly Varma Shrivastav; Anuraag Shrivastav (2017). «Myristoylation: An Important Protein Modification in the Immune Response». Frontiers in Immunology 8: 751.
- ↑ Poveda-Cuevas, Sergio Alejandro; Kateryna Lohachova; Borna Markusic; Ivan Dikic; Gerhard Hummer; Ramachandra M. Bhaskara (2024). «Intrinsically Disordered Region Amplifies Membrane Remodeling to Augment Selective ER-Phagy.». Proceedings of the National Academy of Sciences 121 (44): e2408071121. PMC 11536123. PMID 39453744. (requiere suscripción).
- ↑ Saltar a: a b c d e f g h i j k Mo, Jie; Chen, Jin; Zhang, Bixiang (2020). «Critical Roles of FAM134B in ER-Phagy and Diseases». Cell death & disease (REVISIÓN) 11 (11): 983. PMC 7670425. PMID 33199694.
- ↑ D’Eletto, Manuela; Oliverio, Serafina; Di Sano, Federica (2020). «Reticulon Homology Domain-Containing Proteins and ER-Phagy». Frontiers in Cell and Developmental Biology. (Front Cell Dev Biol) (REVISIÓN) 8 (90). PMC 7047209. PMID 32154249.
- ↑ Knupp, Jeffrey; Madison L. Pletan; Peter Arvan; Billy Tsai (2023). «Autophagy of the ER: The Secretome Finds the Lysosome». The FEBS Journal 290 (24): 5656-5673.
- ↑ Saltar a: a b c Tong, Jia; Qianqian Wang; Ziyan Gao; Yang Liu; Chengbiao Lu (2024). «VMP1: A Multifaceted Regulator of Cellular Homeostasis with Implications in Disease Pathology». Frontiers in Cell and Developmental Biology (REVISIÓN) 12: 1436420.
- ↑ Saltar a: a b Duan, Yu; Ren-qi Yao; Hua Ling; Li-yu Zheng; Qi Fan; Qiong Li; Lu Wang; et al. (2024). «Organellophagy Regulates Cell Death:A Potential Therapeutic Target for Inflammatory Diseases.». Journal of Advanced Research (REVISIÓN): S2090-1232(24)00203-0.
- ↑ Hill, Melissa A.; Sykes, Alex M.; Mellic, George D. (2023). «Er-Phagy in Neurodegeneration». Journal of neuroscience research 101 (10p): 1611-1623.
- ↑ Hübner, Christian A.; Ivan Dikic (2019). «Er-Phagy and Human Diseases». Nature News 27: 833-842.
- ↑ Farhadul Islam; Vinod Gopalan; Alfred King-Yin Lam (2018). «Retreg1 (FAM134B): A New Player in Human Diseases: 15 Years after the Discovery in Cancer». Journal of cellular physiology (REVISIÓN) 233 (6): 4479-4489. PMID 29226326. doi:10.1002/jcp.26384. (requiere suscripción).